Huella genética de los morfotipos del árbol del ramón
Los ensayos han sido realizados con la colaboración del grupo de GemBio del Centro de Investigación Científica de Yucatán. Se ha obtenido un gran avance.
Para continuar con el estudio de la huella genética del Ramón se utilizaron 5 Iniciadores DAMD (Direct). A diferencia de los marcadores ISSR, todos los DAMD mostraron polimorfismo en las muestras analizadas, el análisis de distancia genética se realizó con el programa NTSYSpc 2.21q.
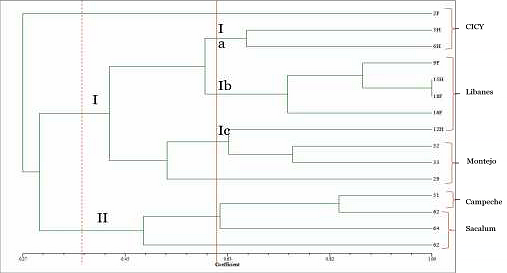
En la figura 1 se pueden ver dos clados bien definidos con una distancia genética de 0.3. En el clado (I) se agrupan todas las muestras de Mérida, mientras que en el (II) se agruparon las muestras de Hopelchén (Campeche) y Sacalum (Yucatán). Al mismo tiempo el clado (I) se divide en 3 subclados, de acuerdo a la zona de muestreo, con una menor diferencia genética entre ellos: (Ia) muestras provenientes del CICY, (Ib) muestras del Libanés y el (Ic) las muestras de Paseo de Montejo. Hasta el momento los marcadores DAMD muestran una mayor resolución de agrupamiento de acuerdo a la zona de muestreo, lo cual se traduce en una alta variabilidad intra-específica en Brosimum alicastrum.
Figura 1. Dendograma obtenido con UPGMA, basado en el análisis de DAMD con el iniciador M13.
Fuente: elaboración propia.
Actualmente se está realizando el análisis de los parámetros genéticos por cada iniciador de ISSR y DAMD, así como el análisis combinado de datos entre los dos tipos de marcador.
Con el objetivo de realizar la caracterización molecular de Brosimum alicastrum se realizo un muestreo de hojas de árboles de Ramón los cuales presentaban características muy particulares y que entre ellos mismos mostraban diferencias en su morfología, en diferentes localidades de Yucatán y Campeche (Izamal; Sacalum; Abala; Mérida y Hopelchén. Se colectaron hojas de 25 árboles de ramón con diferencia en el sexo y su morfología pertenecientes a 4 zonas de la ciudad de Mérida (cuadro 1) para analizarlas molecularmente a través de la técnica de inter-secuencias simples repetidas (ISSR).
Cuadro 1. Colecta de muestras de Ramón
| Clave muestra | Ubicación | Clave muestra | Ubicación | Clave muestra | Ubicación |
|---|---|---|---|---|---|
| R1 | Salón líbano | R11 | Jardín botánico CICY | R21 | Av. Paseo de Montejo |
| R2 | Salón líbano | R12 | Jardín botánico CICY | R22 | Av. Paseo de Montejo |
| R3 | Salón líbano | R13 | Jardín botánico CICY | R23 | Av. Paseo de Montejo |
| R4 | Salón líbano | R14 | Jardín botánico CICY | R24 | Itzimná |
| R5 | Salón líbano | R15 | Jardín botánico CICY | R25 | Itzimná |
| R6 | Salón líbano | R16 | Jardín botánico CICY | ||
| R7 | Salón líbano | R17 | Jardín botánico CICY | ||
| R8 | Salón líbano | R18 | Jardín botánico CICY | ||
| R9 | Salón líbano | R19 | Jardín botánico CICY | ||
| R10 | Salón líbano | R20 | Jardín botánico CICY |
Fuente: elaboración propia.
La extracción de ADN se realizó mediante el protocolo descrito por Tapia-Tussell et al. (2005). Se verificó la integridad del ADN extraído así como la concentración y calidad. La concentración estuvo en un rango de 249 a 1115.6 ng/µl, mientras que la pureza fue de 1.3 a 1.8
Para el estudio de la huella genética del Ramón se utilizaron 14 iniciadores ISSR de los cuales solo 7 mostraron polimorfismo en las muestras analizadas, el análisis de distancia genética se realizó con el programa NTSYSpc 2.21q.
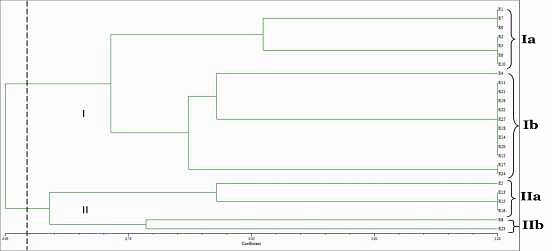
Como se puede apreciar en el dendograma generado con el iniciador IS09 (figura 1) se forman dos grandes clados (I y II). Del total de muestras analizadas el 76% se agrupan en el clado I y el resto (24%) en el clado II.
El clado I, a su vez se divide en dos subclados (Ia y Ib). En el subclado Ia sólo se agrupan muestras que provienen del salón libanés (70%) a diferencia del subclado Ib en el cual se agruparon el 70% de las
muestras provenientes del CICY y las muestras de las otras dos localidades (Av. Paseo Montejo e Itzimná). Esto nos indica de manera preliminar que se puede encontrar con esta técnica una alta variabilidad genética en la especie de Brosimum alicastrum.
Figura 2. Dendograma obtenido con UPGMA, basado en el análisis de ISSR (iniciador IS09).
Fuente: elaboración propia.
Para continuar con el estudio de la huella genética del Ramón se utilizaron 5 Iniciadores DAMD (Direct ). A diferencia de los marcadores ISSR, todos los DAMD mostraron polimorfismo en las muestras analizadas, el análisis de distancia genética se realizó con el programa NTSYSpc 2.21q.
En la figura 1 se pueden ver dos clados bien definidos con una distancia genética de 0.3. En el clado (I) se agrupan todas las muestras de Mérida, mientras que en el (II) se agruparon las muestras de Hopelchén (Campeche) y Sacalum (Yucatán). Al mismo tiempo el clado (I) se divide en 3 subclados, de acuerdo a la zona de muestreo, con una menor diferencia genética entre ellos: (Ia) muestras provenientes del CICY, (Ib) muestras del Libanés y el (Ic) las muestras de Paseo de Montejo. Hasta el momento los marcadores DAMD muestran una mayor resolución de agrupamiento de acuerdo a la zona de muestreo, lo cual se traduce en una alta variabilidad intra-específica en Brosimum alicastrum.
Actualmente se está realizando el análisis de los parámetros genéticos por cada iniciador de ISSR y DAMD, así como el análisis combinado de datos entre los dos tipos de marcador.